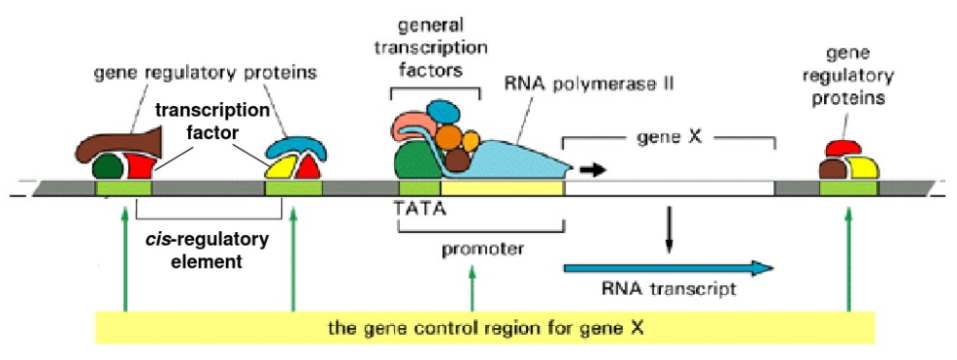
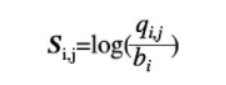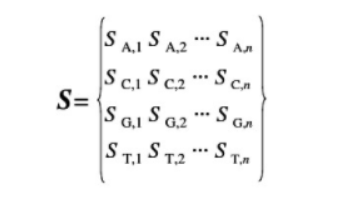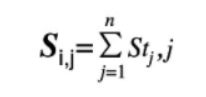MRs是指能够对多个靶基因进行调控的转录因子(TFs),人体基因组大概包含了1800序列特异性的TF。每一个TF可以调节数百个目标基因。大约40%的肿瘤变异基因通过影响TF影响发病机制,一些比较著名的肿瘤基因比如p53、MYC、E2F这些都是转录因子TF。
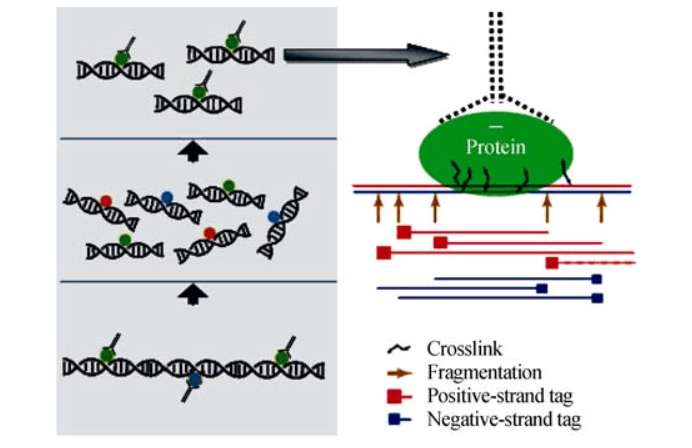
知道转录因子,可用ChIP技术寻找其所有的目标基因,但是ChIP-Seq一次只能做一个TF,而且事先需要知道TF,对于只知道基因表达的情况不太适用。
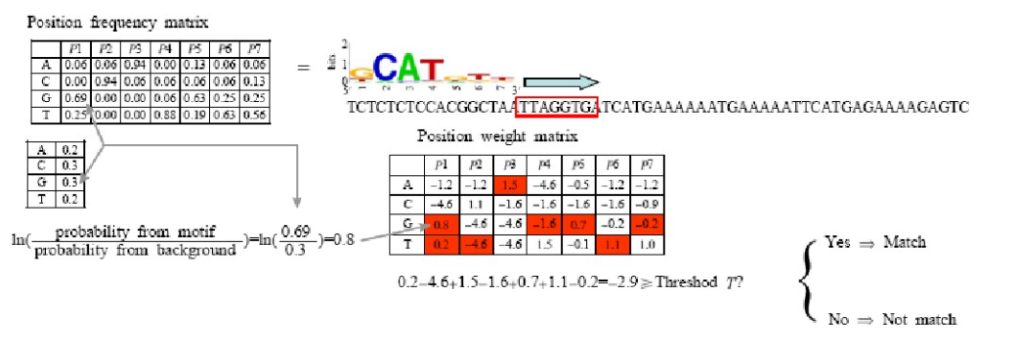
PWM全称是位置权重矩阵,被广泛使用在识别转录因子基序。其基本原理是,受同一转录因子调节的基因具有相似的DNA结合序列。
(一)共有序列
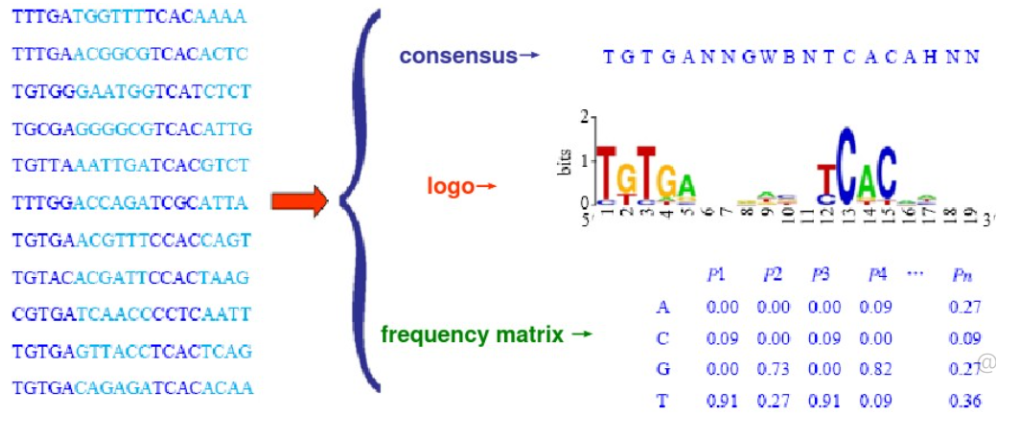
将能与同一个转录因子结合的所有DNA片段按照对应位置进行排列,在每个位置上选择最有可能出现的碱基,就组成了该转录因子结合位点的共有序列。可以使用序列标识图来表示。
(二)位置频率矩阵
位置频率矩阵可以反映出每个位置上不同碱基出现的概率,该模型的一个前提假设是各个位置上碱基出现的概率相互独立,矩阵每一列表示模体相应位置上四种碱基出现的概率。
(三)位置权重矩阵
对任一长度为n的已知模体位置频率矩阵M,转录因子结合位点定位就是判断某一长度为n的序列片段与M的匹配程度。考虑到DNA序列本身可能存在碱基组成上的偏向性,通常把位置频率矩阵转换成位置权重矩阵。用位置权重矩阵的打分来衡量模体与任意给定序列的匹配程度。
(1)在位置权重矩阵中,为了消除DNA序列本身碱基组成偏好性的影响,所以引入了碱基i(i={A,T,C,G})在背景序列中出现的频率(记为bi)
(2)位置频率矩阵M被转换为位置权重矩阵
(3)对于长度为n的DNA序列片段,它作为模体M对应的转录因子结合位点的打分为
(4)给定一个基因序列,利用位置权重矩阵寻找转录因子可能的结合位点。
iRegulon是Cytoscape的一个插件,能够支持人、鼠、果蝇基因的富集。当我们有一些表达差异的基因,iRegulon能够告诉我们其中的master调节因子是什么。当我们想要知道一个已知的TF其调控的下游基因是什么,也可以使用iRegulon。
论文链接:https://www.ncbi.nlm.nih.gov/pmc/articles/PMC4109854/
GitHub链接:https://github.com/aertslab/iRegulon
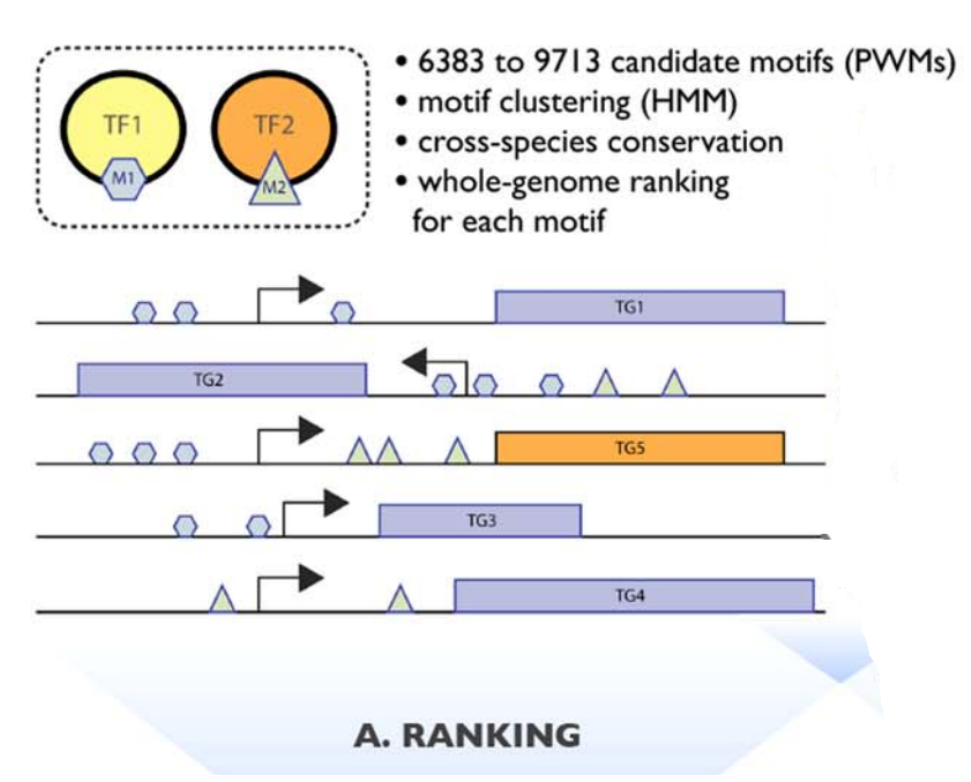
从各种数据库中收集到9000多个PWM和1000多个ChIP-Seq数据,而且这些PWM都对应到了相应的TF上,每个TF可能对应多个PWM。
(1)排序,对22284个人基因进行排序,排序的依据就是PWM库,有两种PWM库,一个是6K库,一个是10K库,主要包含的PWM数量不同。一个PWM库就是一个motif 的量化表征,对每个基因来说,搜寻其转录起始点附近的区域的CRM(顺式调控元件),然后对其进行打分,根据分数高低不同进行排序。最终结果就是一个SQLite数据库,这个数据库中包含有N个基因的排序。
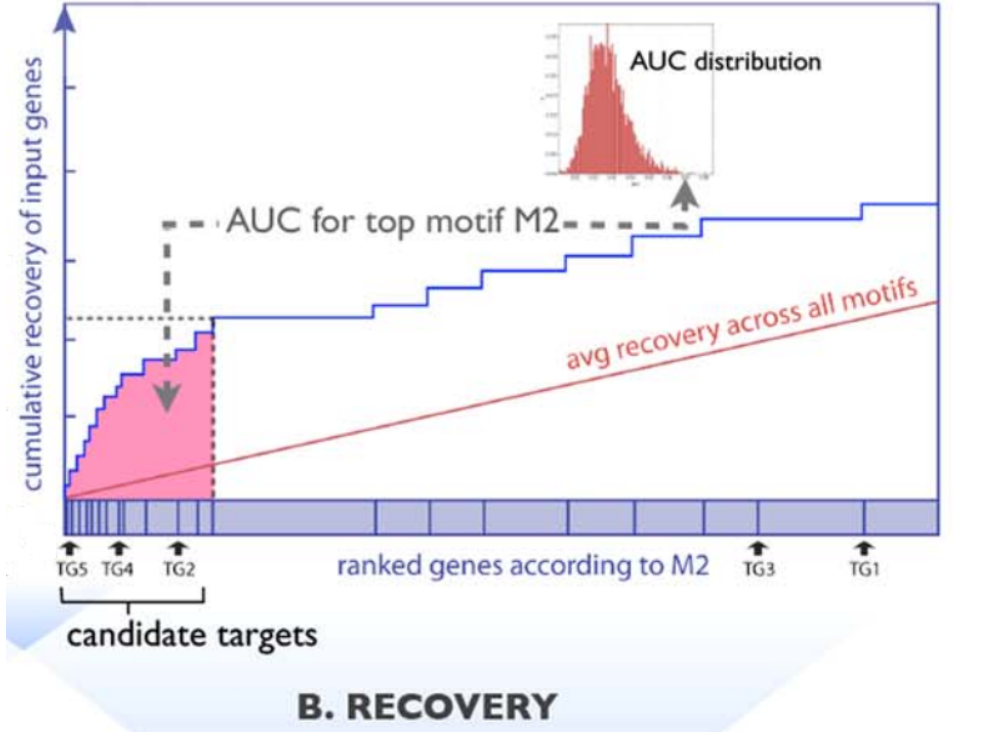
(2)回收,需要共表达基因作为输入信息。就是探讨这些基因在哪些PWM所对应的基因排序中富集,可以用累计回收曲线下面积(AUC)表示。AUC通过计算前3%基因里包含多少输入的共表达基因。前3%基因就是22284 × 0.03 = 669个基因。假如,一共输入了100个共表达基因,其中80个在前669个基因里,AUC = 80/100 = 0.8,说明在排序3%回收了80%的基因,这时候就可以认为这个PWM所对应的TF就是一个Master Regulators。
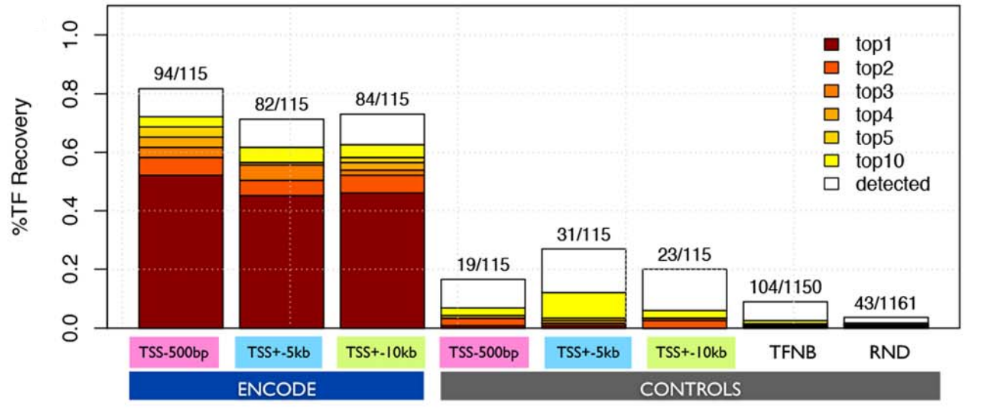
(1)使用115个序列特异性的TF,这些TF的下游目标基因都是已知的,可以从现有的数据库中获得。验证就是将目标基因集输入iRegulon,返回的TF(根据归一化富集评分对返回的TF进行排序,从而得出top1、top2、top3)与已知的TF进行比较。iRegulon能将82.6%的TF正确识别。Controls使用的是阴性基因集。
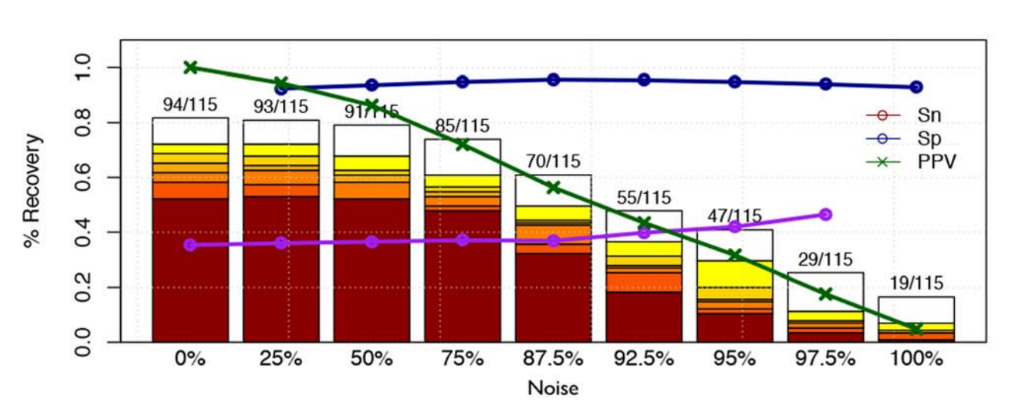
(2)观察噪声(阴性基因)对模型的影响,随着噪声的增加,TF回收率不断减少。sn是指灵敏度,sp是指特异性,ppv是指精度。
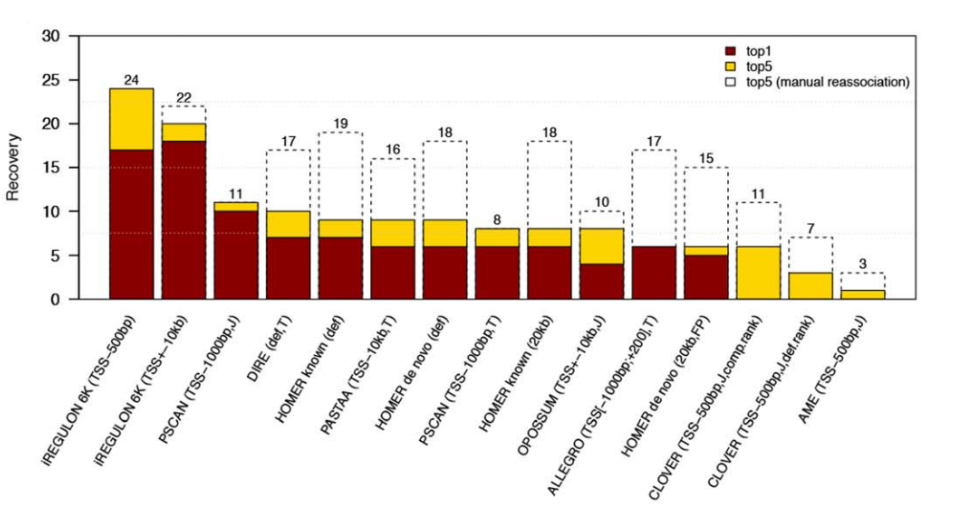
(3)与其他预测工具比较,使用30个TF,每个TF选取通过ChIP获得的前200个目标基因。
(1)下载地址:https://cytoscape.org/download.html
安装之后,打开软件的界面。
(2)安装iRegulon插件
1)打开Apps的Apps Manager
2)在Search搜索插件iRegulon,然后install
3)安装成功后
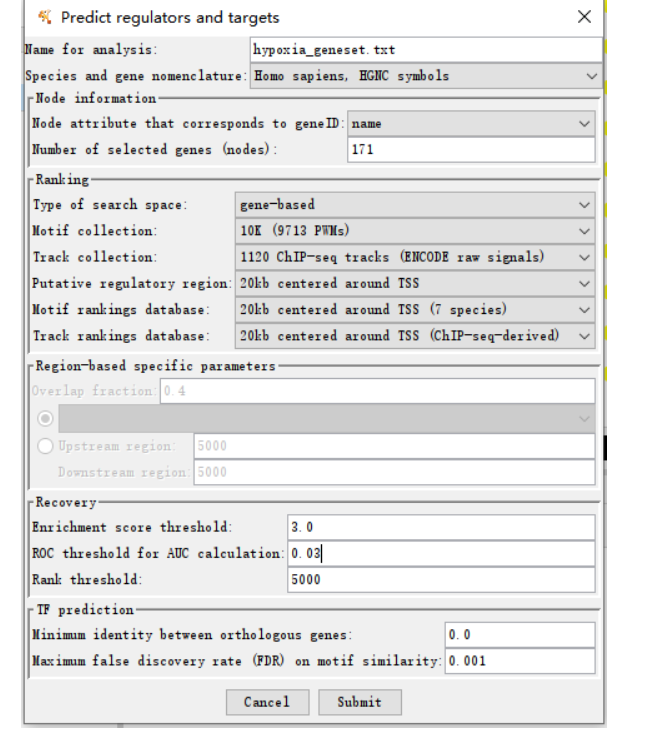
(1)整理好需要输入共表达的基因,这里整理的是跟缺氧有关的171个共表达基因
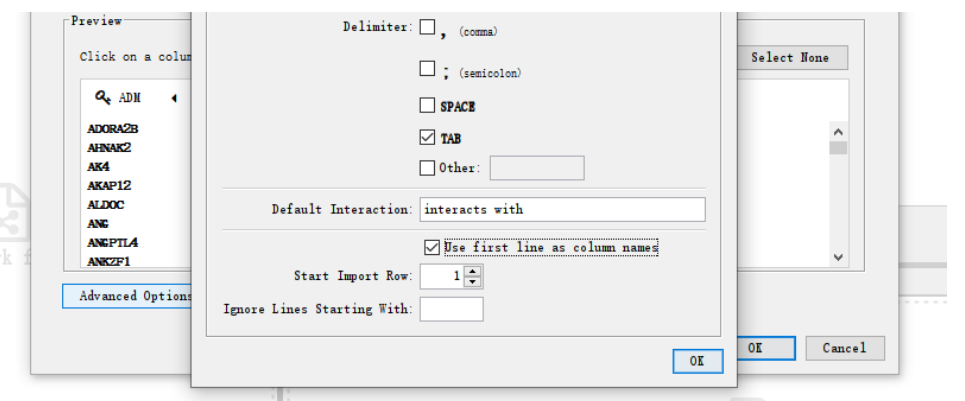
(2)导入文件
(3)取消使用第一行作为行名
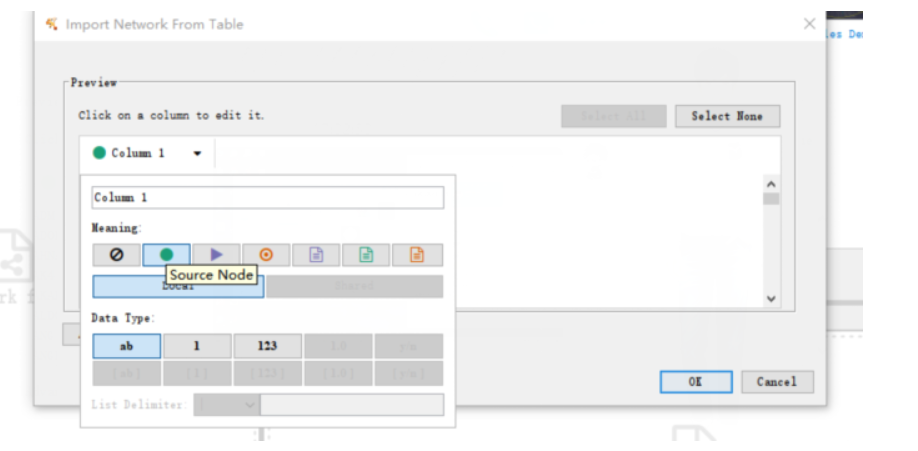
(4)点开column1,选择Source Node
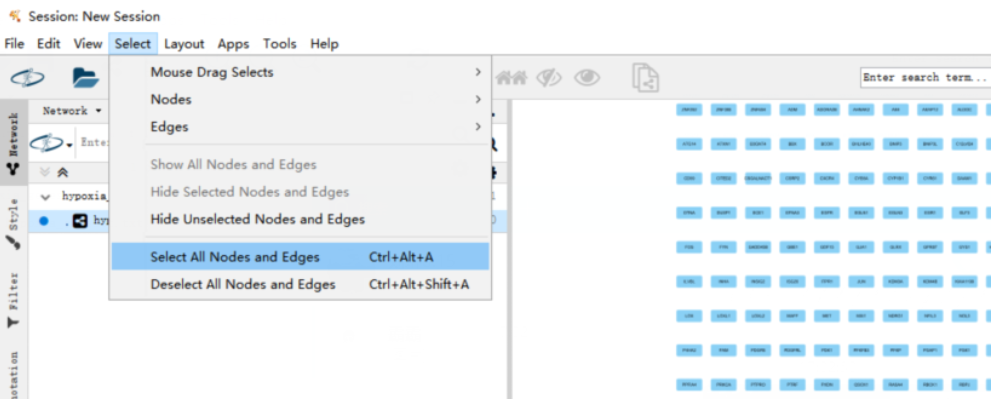
(5)选择所有nodes 和 edges
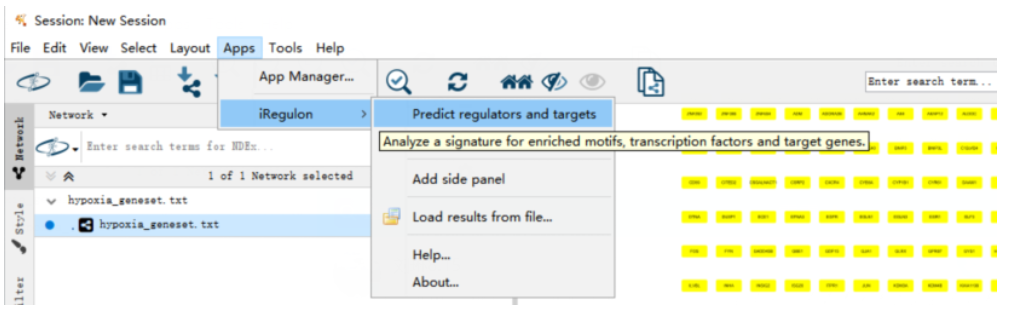
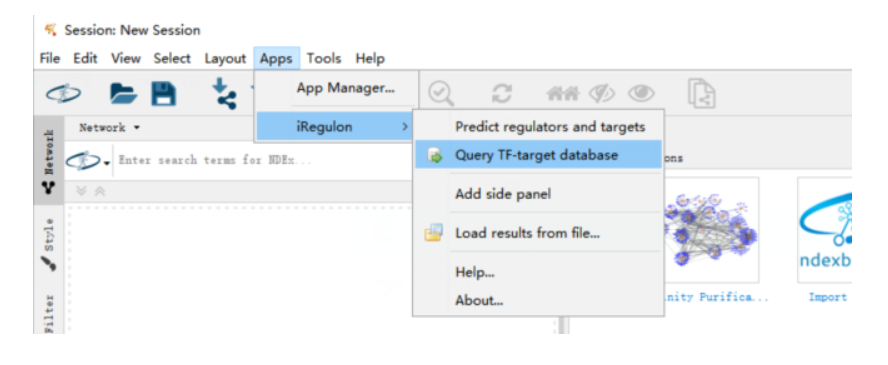
(6)使用iRegulon插件,选择Apps中的预测regulators和targets
(7)进行参数设定。
species可以选择智人、小鼠 和果蝇。
Motif cllection可以选择10K(9713个PWMs),或者6K(6383个PWMs)
Track cllection可以选择1120个ChIP-seq(raw signals),或者750个ChIP-seq(uniform signals)
Putative regulatory region可以选择20kb、10kb、500bp
ROC threshold for AUC calculation默认设定是3%
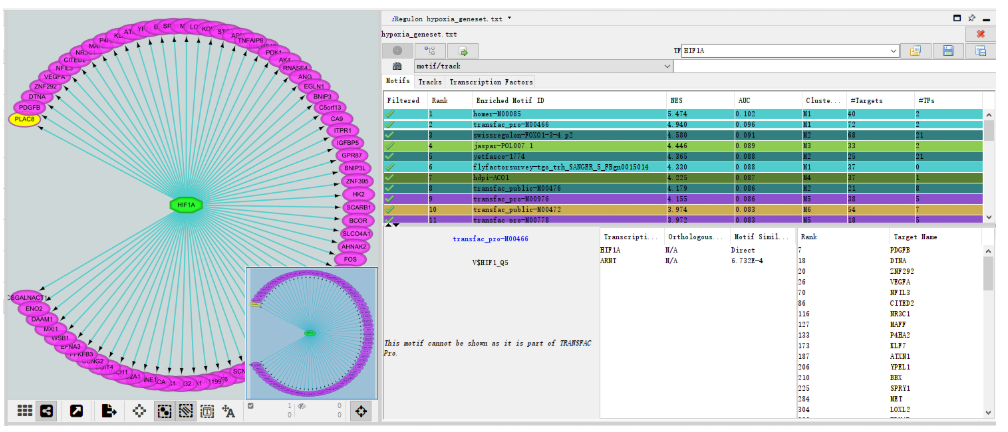
(8)结果。NES评分为4.940,转录因子HIF1A和ARNT富集到了72个目标基因,
(1)使用iRegulon插件
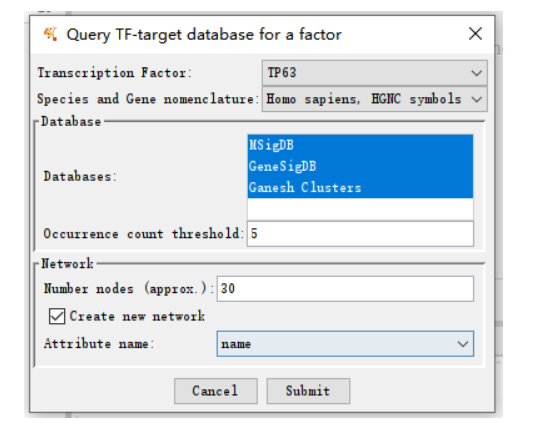
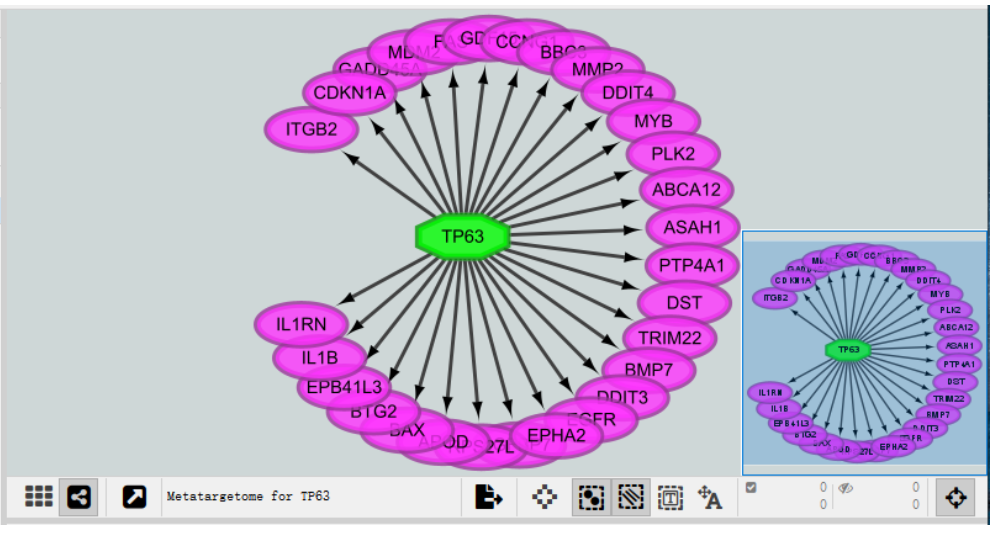
(2)以转录因子TP63为例,可以选择一个数据库,也可以同时选择多个数据库。
(3)结果展示